“
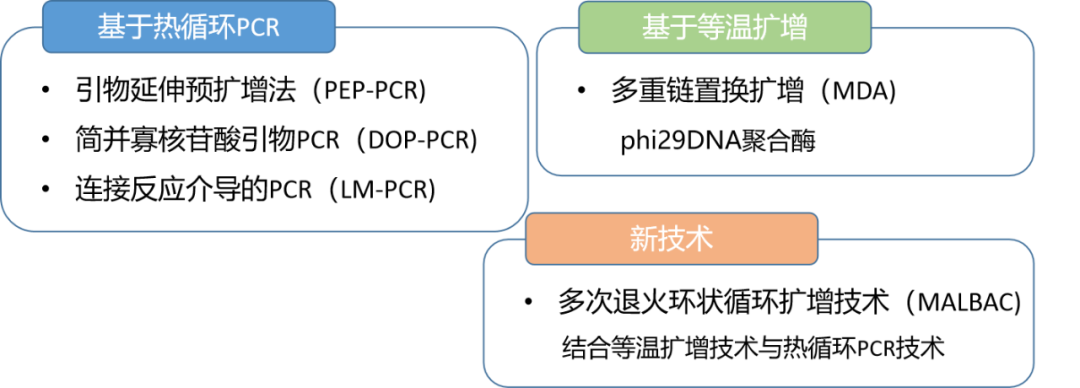
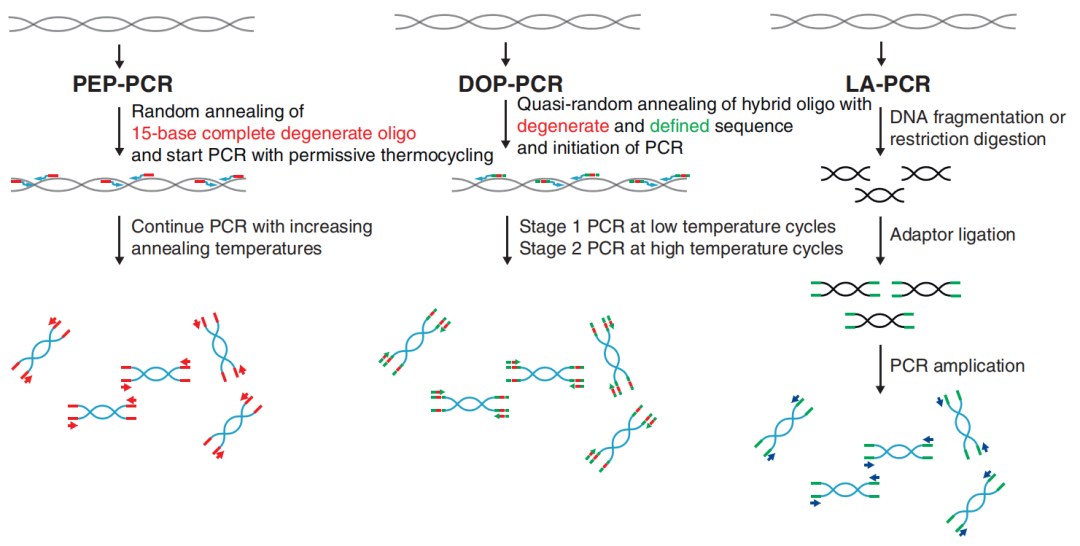
PEP-PCR(primer extension preamplification PCR)
“
DOP-PCR(degenerate oligonucleotide primed PCR)
“
LM-PCR(ligation-mediated PCR)
“
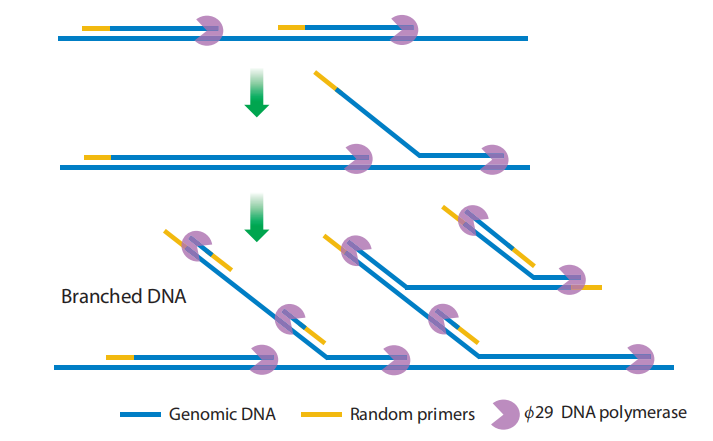
多重置換擴(kuò)增技術(shù)MDA(multiple displacement amplification)
“
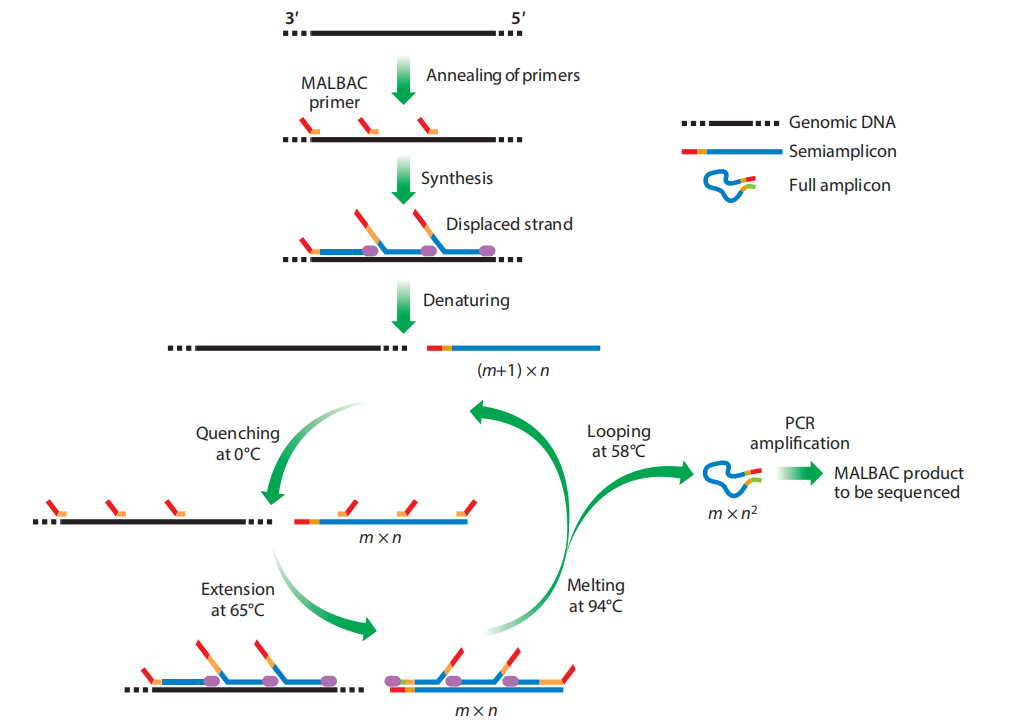
MALBAC(multiple annealing and looping-based amplification cycles)